近日,作物病原生物功能基因组研究创新团队在国际知名期刊《基因组生物学》(Genome Biology)上在线发表研究论文“A large-scale genome and transcriptome sequencing analysis reveals the mutation landscapes induced by high-activity adenine base editors in plants”,该研究利用基因组和转录组测序数据系统分析了团队开发的高效腺嘌呤碱基编辑器(ABE,Mol Plant, 2021, 14: 722-731)在DNA和RNA水平上的脱靶效应,为提高ABE碱基编辑特异性提供了新策略。

CRISPR碱基编辑工具可以在基因组指定位置上进行A>G (腺嘌呤碱基编辑器ABE)或者C>T(胞嘧啶碱基编辑器CBE)的突变,因此被广泛应用于动植物育种、基因治疗和基础科学研究中,而脱靶效应是限制其应用的关键因素。鉴于水稻相对较小的基因组,自花授粉的遗传特征,以及缺少ADAR基因家族(介导RNA水平上的A>I 突变),水稻是研究CRISPR碱基编辑工具特异性的模式生物。
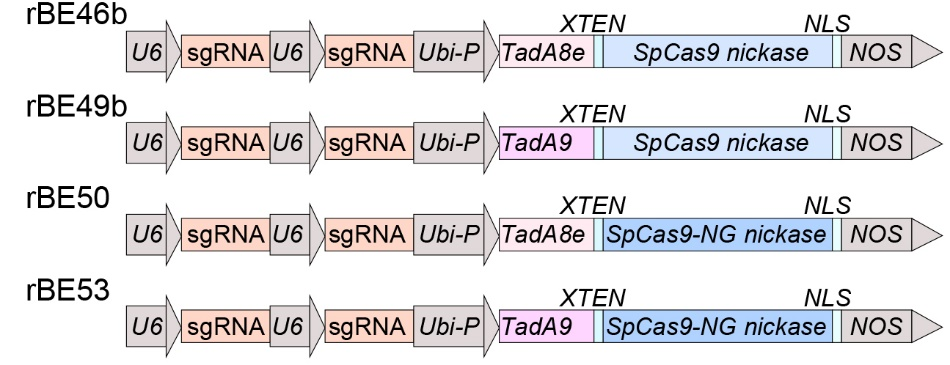
腺嘌呤碱基编辑器ABE含有CRISPR和腺苷脱氨酶(TadA)两个重要组分 :SpCas9n-NG和SpCas9n是两个适用性最高的CRISPR组分,TadA8和TadA9是两个具有普适性的高效腺苷脱氨酶。但是TadA8e和TadA9在基因组范围内引起的DNA脱靶效应还未被评估,此外,TadA引起植物RNA的脱靶效应也未被报道。在前期研究中,该团队开发了四套含有这几个组分的ABE工具:SpCas9n-TadA8e, SpCas9n-TadA9, SpCas9n-NG-TadA8e和SpCas9n-NG-TadA9。在此项研究中,研究人员对转化这四套ABE的水稻基因编辑植株进行了全基因组和转录组测序并进行了脱靶效应的系统分析。
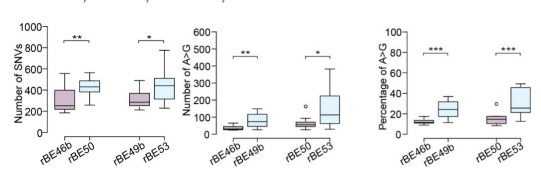
ABE可引起sgRNA依赖和sgRNA非依赖的脱靶效应,该研究发现含有优化sgRNA的ABE并不引发sgRNA依赖的脱靶效应。而CRSPR和TadA两个组分都会影响ABE在DNA水平上sgRNA非依赖的脱靶效应。SpCas9n-NG 比SpCas9n识别更灵活的PAM结构,携带SpCas9n-NG的ABE造成了更多总体水平上单核苷酸突变(SNVs)。TadA9 比TadA8e的活性更高,编辑窗口更宽,携带SpCas9n-NG的ABE造成了更多A>G的SNVs。在转化SpCas9n-TadA8e的水稻植物中,研究人员并未检测到ABE引起的脱靶突变。此外,本研究通过生信分析证明ABE引起的sgRNA非依赖性、的脱靶位点是完全随机的。
动物中特有的ADAR家族可引起mRNA产生A>I突变,并以A>G的形式存在于转录组数据中,而水稻植株中缺少ADAR家族。此项研究首次报道了TadA在RNA水平上引起序列依赖性的A>G脱靶效应,ABE表达水平是影响其脱靶效应的重要因素。当ABE从T1代水稻植株分离后,ABE在RNA上的脱靶效应随之消失。此项研究还发现ABE在RNA的脱靶突变往往成簇存在,而DNA水平的脱靶突变则很少成簇出现。
农杆菌介导的T-DNA是植物中传递基因编辑工具的主要方式,此研究发现ABE在整合到植物基因组之前和之后均可以引起靶位点碱基编辑。来源于同一转化事件的不同水稻植株含有相同T-DNA插入位点,而靶位点的碱基编辑却可能不同,同时这些水稻植株之间却含有一部分相同的脱靶位点,因此选取两株以上不同转化事件的植株是避免脱靶效应产生假阳性的重要手段。
概言之,此项研究通过系统分析基因组和转录组数据,首次报道了高效ABE8e和ABE9在DNA水平上的脱靶效应,首次报道了ABE在植物个体中引起的RNA水平上的脱靶效应。研究人员发现TadA、CRISPR、ABE的瞬时表达以及表达水平都会影响ABE的特异性,这一研究成果也为ABE的优化指明了新方向。
该研究得到国家自然科学基金和中央公益性科研机构基础研究基金项目资助。原文链接:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-022-02618-w