植保所农业入侵生物预防与监控创新团队和南繁院南繁生物安全与风险评估团队采用了Nanopore长读序列、Illumina短读序列和染色体构象捕获(Hi-C)技术相结合的方法,首次将广聚萤叶甲 Ophraella communa 参考基因组提升至染色体水平,进行了结构和功能注释。该基因组为萤叶甲属中首个染色体水平的基因组组装,这一高质量的参考基因组不仅为更好地提升广聚萤叶甲的生物防治潜力提供了可靠的基因组数据基础,也为理解萤叶甲属的遗传学、生态学和系统进化提供宝贵的数据资源,研究结果于2024年6月在线发表于SCI刊物 Scientific Data 。
广聚萤叶甲原产于北美,为入侵杂草豚草 Ambrosia artemisiifolia 的专一性天敌昆虫,对豚草非常好的控制效果。为了更好地应用广聚萤叶甲,其化学生态学、繁殖生物学和耐寒遗传学的研究一直在进行中,原有的基因组草图已不能满足目前研究的需求。
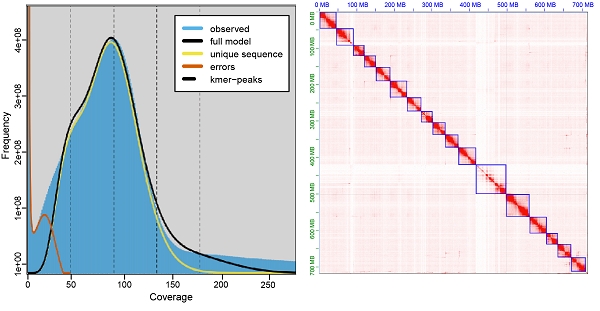
研究人员首先构建了广聚萤叶甲的实验室种群,经过多代近交降低杂合度后,测序获得Nanopore长读长序列和Illumina短读长序列,分别用于组装和矫正基因组,得到基因组大小为735.31 Mb,contig N50为7.05 Mb的组装结果。结合染色体构象捕获(Hi-C)技术,将基因组挂载到17条染色体上。结构注释共鉴定了25,873个蛋白质编码基因,其中22,084个基因同时被功能注释。此外,在基因组中注释了204个 rRNA,626个 tRNA,1791个小 RNA。重复序列注释得到的重复元件占基因组的414.41 Mb (57.76%)。BUSCO评估基因组组装完整度为99.7%,注释完整度为95.1%,利用Illumina短读长序列对基因组组装的准确性进行评估,比对率为99.56%。
中国农业科学院植保所在读博士生王奕婷和博士后张燕为共同第一作者;中国农业科学院植物保护研究所周忠实研究员和北京市农林科学院植保所魏书军研究员为论文通讯作者;中国农业科学院植物保护研究所马超博士、华中农业大学植物科学技术学院马伟华教授、北京市农林科学院植保所曹利军副研究员、陈金翠助理研究员、宋威博士后、中国农业科学院植物保护研究所杨景芳助理研究员、广西农业科学院植物保护研究所高旭渊副研究员和陈红松研究员、中国农业科学院植保所田震亚博士,法国国家农业研究院Nicolas Desneux研究员,参与了部分工作。该研究得到国家重点研发项目(项目号:2023YFE0104800、2022YFC2601400)、国家自然科学基金项目(项目号:32172494)以及北京市农林科学院项目(项目号:JKZX202208)的支持。